Essa é uma revisão anterior do documento!
Tabela de conteúdos
José Cláudio Faria
Eu na Praia do Sul de Ilhéus/BA, em janeiro de 2007, refletindo profundamente sobre estatística computacional e o R!!!
Brincadeiras a parte…
1. Quem sou
- Engenheiro Agrônomo;
- Mestrado e Doutorado em Produção Vegetal pela Universidade Federal de Viçosa - UFV/MG.
2. O que tenho feito profissionalmente
- Professor de estatística e pesquisador da Universidade Estadual de Santa Cruz - UESC/BA;
- Coordenador e desenvolvedor do projeto Tinn-R (GUI/editor para o ambiente R).
3. Sobre o R
- Gostaria de tê-lo encontrado desde o início de minha carreira na área de estatística computacional.
4. Sobre o futuro
- Desejo aprofundar os conhecimentos em análise multivariada de dados no ambiente R;
- Aprimorar o Tinn-R e disponibilizá-lo também para a plataforma Linux;
- Trocar experiências com pessoas e equipes envolvidas nestas áreas.
Tinn-R
Tinn-R GUI/Editor para o ambiente R sob Windows.
- O Tinn-R é um programa de código aberto (sob GPL) desenvolvido em Object Pascal com a IDE Delphi_7 da Borland;
- Facilita o uso do interpretador R e aumenta a produtividade das análises e documentações;
- Suporte ao Sweave;
- Permite interagir com o R em modo gráfico, o que aumenta a produtividade e facilita o uso, ao mesmo tempo em que estimula o aprendizado da linguagem R;
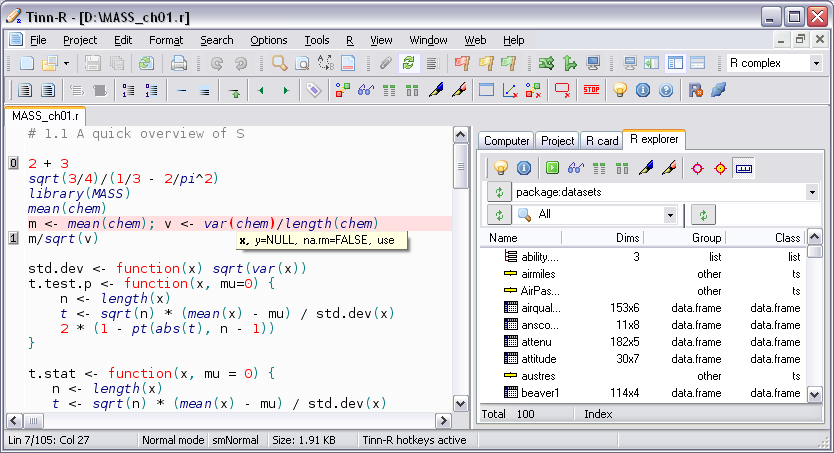
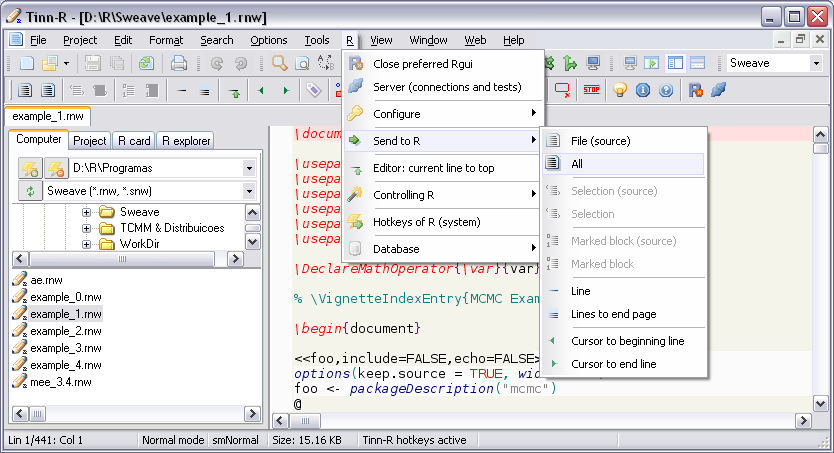
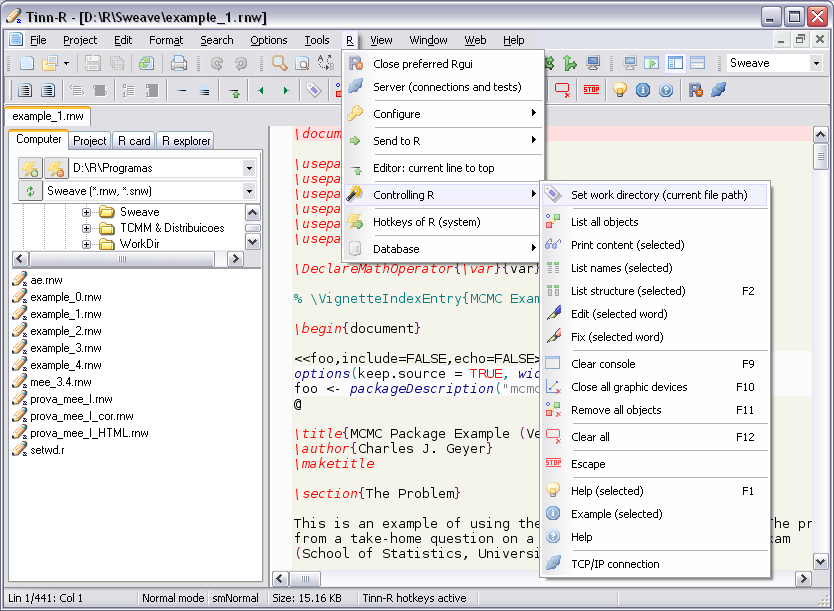
- Imagens:
Materiais sobre o R
Scripts
Todos os usuários estão automaticamente convidados a darem sugestões e alterarem contrutivamente todas as funções e scripts desta página. Solicito a gentileza de me enviar um email comunicando as alterações.
Introdução ao R
Abrir no Tinn-R e executar linha por linha buscando entender cada passo:
#===============================================================================
# Título: Introdução ao R - IR
# Curso : Métodos estatísticos aplicados à produção vegetal
# Autor : José Cláudio Faria/UESC/DCET
# Data : 15/12/06 18:39:16
# Versão: v7 - com comentários - cc
# Objetivos:
#===============================================================================
# a) Apresentar os recursos gráficos básicos do R
# b) Documentação e ajuda
# c) Funções elementares
# d) Estruturas de dados
# e) Operadores
# f) Estruturas de controle de fluxo
# g) Funções
#===============================================================================
#===============================================================================
# Exemplos
#===============================================================================
demo()
demo(package = .packages(all.available = TRUE))
demo(graphics) # Recursos gráficos genéricos
# Para teclar <Enter> to see next plot:
# é necessário que a tela esteja ativa
demo(image) # Recursos gráficos 2D
demo(persp) # Recursos gráficos 3D
library(lattice)
demo(lattice) # Recursos gráficos
demo(glm.vr) # Método lineares generalizados
demo(lm.glm) # Lineares e lineares generalizados
#===============================================================================
# Documentação e ajuda
#===============================================================================
?round
?'for' # ou ?”for“
?'[[' # ou ?”[[“
apropos('stem')
help.search('stem')
help.start() # ou menu 'Help/Html help
vignette() # documentos em pdf (dependente dos pacotes instalados)
vignette('grid') # abre pdf relacionado ao pacote grid
#===============================================================================
# Algumas funções elementares
#===============================================================================
set.seed(25)
x = round(runif(n = 20, min = 0, max = 10), digits = 2)
x
sort(x)
min(x)
max(x)
median(x) # mediana
mean(x) # média
var(x) # variância
sd(x) # desvio padrão (standard deviation)
sqrt(var(x))
sum(x) # somatório
length(x) # número de elementos
round(x, digits = 1)
round(x)
fivenum(x) # Returns Tukey's five number summary (minimum, lower-hinge, median, upper-hinge, maximum)
quantile(x) # quantis
quantile(x, c(0, .33, .66, 1))
cummax(x)
cummin(x)
plot(x, sin(x/20))
cor(x, sin(x/20))
# Imprimir no console uma mensagem ou o valor de uma variável:
print('Teste:')
x = 10
print(x)
# Concatenação:
cat('\nValor de x =', x); cat('\n')
cat('\n\tValor de x =', x); cat('\n')
#===============================================================================
# Estruturas de dados: MUITO IMPORTANTE!!!
#===============================================================================
#===============
# Vetores
#===============
# Algumas das diversas maneiras de defini-los:
c(1, 2, 3, 4, 5)
1:6
seq(1, 10, by = 1)
seq(1, 2, length = 10)
letters[1:5]
LETTERS[1:5]
# Algumas maneiras de recuperá-los:
x = seq(1, 10, by = 1)
x
x[5:10]
x[c(5, 7:10)]
x[-(5:10)]
x > 5
x[x > 5]
x[x < 6]
# Dar nomes aos componentes de um vetor:
names(x)
names(x) = letters[1:length(x)]
x
x['b']
c(a = 1, b = 5, c = 10)
# Algumas operações básicas:
set.seed(3)
x = round(runif(5, 0, 10), d = 1)
x
x/2
x*2
x+10
sort(x)
rev(sort(x))
set.seed(16)
x = sample(1:5, 10, replace = T)
x
sort(x)
unique(x)
x = c(1, 3, 2, 8, 5)
x
o = order(x)
o
x
x[o[1:3]]
#===============
# Matrizes
#===============
m = matrix(c(1, 2, 3, 4), nrow = 2)
m
m[1,2]
# O produto matricial:
x = matrix(c(6, 7), nrow = 2)
x
m %*% x
# O determinante de uma matriz:
det(m)
# A transposta de uma matriz:
t(m)
# Uma matriz diagonal:
diag(c(1,2))
# A identidade da matriz:
diag(1, 2)
diag(rep(1, 2))
diag(2)
# Comandos cbind e o rbind para criar matrizes:
cbind(c(1, 2), c(3, 4))
rbind(c(1, 3), c(2, 4))
# O traço de uma matriz:
sum(diag(m))
# A inversa de uma matriz :
solve(m)
solve(m, x)
solve(m) %*% x
# Autovalores:
eigen(m)$values
# Autovetores:
eigen(m)$vectors
# Certificar se a matriz é realmente diagonalisável:
p = eigen(m)$vectors
d = diag(eigen(m)$values)
p %*% d %*% solve(p)
#===============
# Arrays
#===============
ar = array(letters[1:24], c(2,4,3))
ar
ar[1,1,1] # ar[linha, coluna, dimensão] -> ar(x, y, z)
ar[1,1,2]
ar[1,2,3]
class(iris3)
iris3
#===============
# Fatores
#===============
set.seed(218)
x = factor(sample(c('a', 'b', 'c'), 5, replace = T))
x
l = c('d', 'e', 'f')
l
set.seed(17)
x = factor(sample(l, 5, replace = T), levels = l)
x
levels(x)
# Pode-se preferir uma tabela:
table(x)
# Se os valores estão de acordo com alguma razão, pode-se gerar níveis:
gl(1, 4)
gl(2, 4)
gl(2, 4, labels = c(T, F))
gl(2, 1, 8)
gl(2, 1, 8, labels = c(T, F))
# Pode fazer o produto cartesiano de dois fatores:
x = gl(2, 4)
x
y = gl(2, 1, length = 8)
y
interaction(x, y)
# O comando expand.grid é comparável (ele produz um frame), sendo muito útil para
# geração de níveis de fatores para as matrizes provenientes de dados
# experimentais:
a = c('a1', 'a2', 'a3')
b = c('b1', 'b2')
c = c('c1', 'c2')
dad = expand.grid(a, b, c)
names(dad) = c('A', 'B', 'C')
dad
#===============
# Frames
#===============
n = 10
set.seed(17)
dF = data.frame(x = rnorm(n),
y = sample(c(T, F), n, replace = T))
dF
# O comando str informa (retorna) a estrutura de um objeto e a parte dos dados
# que contém:
str(dF)
# Quando os objetos são armazenados, com sua própria ordem, o comando “unclass”
# da ordem pode alterá-lo:
n = 10
set.seed(3)
x = runif(n)
x
set.seed(19)
y = 1 - 2 * x + rnorm(n)
y
r = lm(y ~ x)
r
str(r)
r$coefficients
r$residuals
summary(r)
# A informação summary sumariza um objeto (aqui, um frame, mas vai bem com
# quase todos objetos):
summary(dF)
dF
# Pode-se ter acesso aos dados das colunas de diversas maneiras:
dF$x
dF[,1]
dF[['x']]
dim(dF)
names(dF)
row.names(dF)
# Ou pode-se mudar o nome das linhas ou das colunas:
names(dF) = c('a', 'b')
row.names(dF) = LETTERS[1:10]
names(dF)
row.names(dF)
str(dF)
# Pode-se ter acesso direto as colunas de um frame usando o comando attach().
# Obs: Não deve esquecer-se de destacá-lo detach() quando terminar:
data(faithful)
str(faithful)
attach(faithful)
str(eruptions)
detach()
#===============
# Listas
#===============
h = list()
h[['foo']] = 1
h[['bar']] = c('a', 'b', 'c')
str(h)
# Por exemplo, os parâmetros gráficos são armazenados em uma lista usada
# como contagens de chopping:
str(par())
h[['bar']] = NULL
str(h)
#===============
# Outros
#===============
# O comando split torna possível separar os dados de acordo com o valor
# de um fator:
n = 10
nn = 100
set.seed(21)
g = factor(round(n * runif(n * nn)))
x = rnorm(n * nn) + sqrt(as.numeric(g))
xg = split(x, g)
boxplot(xg, col = 'lavender', notch = TRUE, varwidth = TRUE)
str(xg)
# O comando apply torna possível aplicar uma função (para o exemplo, a média,
# quais, etc..) a cada coluna (ou linha) de um frame (ou de uma matriz):
options(digits = 4)
set.seed(5)
dF = data.frame(x = rnorm(20),
y = rnorm(20),
z = rnorm(20))
dF
apply(dF, 2, mean)
apply(dF, 2, range)
# Em dimensões mais elevadas:
options(digits=2)
set.seed(2)
m = array(rnorm(10^3), dim = c(10, 10, 10))
a = apply(m, 1, mean)
a
b = apply(m, c(1, 2), mean)
b
apply(b, 1, mean)
# A função tapply permite reagrupar as observações de acordo com o valor dos
# fatores e uma função (média, soma, etc..) para cada grupo obtido assim:
tapply(1:20, gl(2, 10, 20), sum)
by(1:20, gl(2, 10, 20), sum)
# A função sapply aplica a cada elemento de uma lista (ou de um vetor, etc..) e
# se possível retorna um vetor. A função lapply faz a mesma coisa, mas retorna
# uma lista:
x = list(a = rnorm(10),
b = runif(100),
c = rgamma(50, 1))
lapply(x, sd)
sapply(x, sd)
#===============================================================================
# Operadores
#===============================================================================
-5:7
set.seed(3)
x = floor(10*runif(10))
x
x[3]
x[1:3]
x[c(1, 2, 5)]
# O operador $ é reservado para recuperar um elemento de uma lista ou frame:
op = par()
op$col
op[['col']]
a = 'col'
op[[a]]
# A atribuição é feita por <- ou =.
x <- 1.17
x
y = c(1, 2, 3, 4)
y
# O produto de matrizes (% * %):
A = matrix(c(1, 2, 3, 4), nr = 2, nc = 2)
J = matrix(c(1, 0, 2, 1), nr = 2, nc = 2)
A
J
J %x% A
# O operador %o% é usado manufaturar tabelas da multiplicação
# (chama a função exterior com a multiplicação):
A = 1:5
B = 11:15
names(A) = A
names(B) = B
A %o% B
# A divisão euclidiana é %/%, seu restante é %%
1234 %% 3
1234 %/% 3
411*3 + 1
# A sociedade de uma 'unidade' é feita por %in%
17 %in% 1:100
17.1 %in% 1:100
# O operador ~ é usado descrever modelos (ANOVAS, métodos lineares, etc).
# Falaremos sobre ele mais tarde.
# Para mais detalhes (sobre os operadores negligenciados nestas notas)
# consulte o manual:
?'+'
?'<'
?'<-'
?'!'
?'['
?Syntax
?kronecker
?match
library(methods)
?slot
# Pode-se definir seus próprios operadores, pois são função diretas com dois
# argumentos cujo nome começa e as extremidades em %. O seguinte exemplo são
# tração do manual.
'%w/o%' = function(x, y) x[!x %in% y]
(1:10) %w/o% c(3,7,12)
#===============================================================================
# Estruturas de controle
#===============================================================================
set.seed(15)
x = rnorm(10)
x
y = ifelse(x > 0, 1, -1)
y
z = ifelse(x > 0, 1, ifelse(x < 0, '< zero', 0))
z
#===============
# Conexão:
#===============
set.seed(59)
x = letters[floor(1 + runif(1, 0, 4))]
x
y = switch(x,
a='Bonjour',
b='Gutten Tag',
c='Hello',
d='Konnichi wa')
y
#===============
# Loop for:
#===============
a = 0
for (i in 1:20) {
a = i
if(a <= 5 ) {
cat('a = ', a, '(<= 5)'); cat('\n')
next
}
if(a == 18) {
cat('a = ', a, '(= 18)'); cat('\n')
break
}
}
#===============
# Loop while:
#===============
a = 0
while (a < 11) {
if (a >= 3) print(a)
else cat('não\n')
a = a + 1 # expressão avaliada..
}
#===============
# Loop repeat:
#===============
a = 0
repeat {
a = a + 1
if (a >= 3) print(a)
else cat('não\n')
if (a == 10) break
}
#===============================================================================
# Funções
#===============================================================================
f = function(x) x/10 + 1
f(x = 10)
f(10) # Chamada alternativa
f = function(x) {
x/10 + 1
}
f(x = 10)
f(10) # Chamada alternativa
# Pode atribuir valores aos argumentos:
f = function(x, y = 3) {
x/10 + 1 - y
}
f(10)
# Na chamada da função, pode-se usar o nome dos argumentos, passar novos valores
# para as variáveis, não sendo necessário que os mesmos sigam a ordem declarada
# na função (desde que os valores sejam acompanhados dos respectivos nomes):
f(y = 1, x = 10)
f = function(x, y) {
x/10 + 1 - y
}
f(1, 10)
f(10, 1)
# No fim dos argumentos, pode haver três pontos, representando todos os
# argumentos não especificados:
f = function(x, ...) {
plot(x, ...)
}
Funções úteis
Tabelas e histogramas
Função tb.table
Função simples, flexível mas poderosa para descrever, via tabela de distribuição de freqüências e histogramas, vetores e data.frames.
#===============================================================================
# Name : tb.table
# Original author: José Cláudio Faria, Gabor Gothendievisk and Enio Jelihovschi
# Date (dd/mm/yy): 1/3/07 11:06:02
# Version : v24
# Aim : To make tables of frequency distribution and associated
# histogram
#===============================================================================
# Arguments:
# breaks : Method to determine number of classes= c('Sturges', 'Scott', 'FD')
# by : Variable to group
# end : Last class (high value)
# h : Classes extent
# k : Class number
# right : Intervals right open (default = FALSE)
# start : First class (small value)
# x : A R object (vector or data.frame)
# histogram : Plot histogram (default = TRUE)
# title.histogram: Title of histogram c('auto', 'none')
#===============================================================================
# Common functions
tb.make.table.I <- function(x,
start,
end,
h,
right,
histogram,
titleH)
{
f <- table(cut(x, br=seq(start, end, h), right=right)) # Absolut freq
fr <- f/length(x) # Relative freq
frP <- 100*(f/length(x)) # Relative freq, %
fac <- cumsum(f) # Cumulative freq
facP <- 100*(cumsum(f/length(x))) # Cumulative freq, %
fi <- round(f, 2)
fr <- round(as.numeric(fr), 2)
frP <- round(as.numeric(frP), 2)
fac <- round(as.numeric(fac), 2)
facP <- round(as.numeric(facP),2)
res <- data.frame(fi, fr, frP, fac, facP) # Make final table
names(res) <- c('Class limits', 'fi', 'fr', 'fr(%)', 'fac', 'fac(%)')
# Making the histogram: With Benilton suggestions
if (histogram) {
hist(x,
breaks = seq(start, end, h),
freq = T,
right = right,
xlab = 'Class limits', ylab='Frequency',
col = 'LightYellow',
main = titleH,
xlim = c(start, end), ylim=c(0, max(fi)),
las = 1,
xaxt = 'n')
axis(1, at=round(seq(start, end, h), 2))
}
return(res)
}
tb.make.table.II <- function (x,
k,
breaks=c('Sturges', 'Scott', 'FD'),
right=FALSE,
histogram,
titleH)
{
x <- na.omit(x)
# User defines only x and/or 'breaks'
# (x, {k,}[breaks, right])
if (missing(k)) {
brk <- match.arg(breaks)
switch(brk,
Sturges = k <- nclass.Sturges(x),
Scott = k <- nclass.scott(x),
FD = k <- nclass.FD(x))
tmp <- range(x)
start <- tmp[1] - abs(tmp[2])/100
end <- tmp[2] + abs(tmp[2])/100
R <- end-start
h <- R/k
}
# User defines 'x' and 'k'
# (x, k,[breaks, right])
else {
tmp <- range(x)
start <- tmp[1] - abs(tmp[2])/100
end <- tmp[2] + abs(tmp[2])/100
R <- end-start
h <- R/abs(k)
}
tbl <- tb.make.table.I(x, start, end, h, right, histogram, titleH)
return(tbl)
}
# With Gabor Grotendieck suggestions (thanks Gabor, very much!)
tb.table <- function(x, ...) UseMethod("tb.table")
# Table form vectors
tb.table.default <- function(x,
k,
start,
end,
h,
breaks=c('Sturges', 'Scott', 'FD'),
right=FALSE,
histogram=TRUE,
title.histogram=c('auto', 'none'))
{
# User defines nothing or not 'x' isn't numeric -> stop
stopifnot(is.numeric(x))
x <- na.omit(x)
# User defines only 'x'
# (x, {k, start, end, h}, [breaks, right])
if (missing(k) && missing(start) && missing(end) && missing(h) ) {
brk <- match.arg(breaks)
switch(brk,
Sturges = k <- nclass.Sturges(x),
Scott = k <- nclass.scott(x),
FD = k <- nclass.FD(x))
tmp <- range(x)
start <- tmp[1] - abs(tmp[2])/100
end <- tmp[2] + abs(tmp[2])/100
R <- end-start
h <- R/k
}
# User defines 'x' and 'k'
# (x, k, {start, end, h}, [breaks, right])
else if (missing(start) && missing(end) && missing(h)) {
stopifnot(length(k) >= 1)
tmp <- range(x)
start <- tmp[1] - abs(tmp[2])/100
end <- tmp[2] + abs(tmp[2])/100
R <- end-start
h <- R/abs(k)
}
# User defines 'x', 'start' and 'end'
# (x, {k,} start, end, {h,} [breaks, right])
else if (missing(k) && missing(h)) {
stopifnot(length(start) >= 1, length(end) >=1)
tmp <- range(x)
R <- end-start
k <- sqrt(abs(R))
if (k < 5) k <- 5 # min value of k
h <- R/k
}
# User defines 'x', 'start', 'end' and 'h'
# (x, {k,} start, end, h, [breaks, right])
else if (missing(k)) {
stopifnot(length(start) >= 1, length(end) >= 1, length(h) >= 1)
}
else stop('Please, see the function sintax!')
if (histogram) {
x11()
par(mfrow=c(1, 1))
title.histogram <- match.arg(title.histogram)
switch(title.histogram,
auto = titleH <- 'x',
none = titleH <- '')
}
tbl <- tb.make.table.I(x, start, end, h, right, histogram, titleH)
return(tbl)
}
# Table form data.frames
tb.table.data.frame <- function(df,
k,
by,
breaks=c('Sturges', 'Scott', 'FD'),
right=FALSE,
histogram=TRUE,
title.histogram=c('auto', 'none'))
{
stopifnot(is.data.frame(df))
tmpList <- list()
nameF <- character()
nameY <- character()
# User didn't defines a factor
if (missing(by)) {
logCol <- sapply(df, is.numeric)
nHist <- length(logCol[logCol])
if (histogram) {
count = 0
if (nHist > 1) {
x11()
par(mfrow=c(4, 1))
}
}
for (i in 1:ncol(df)) {
if (logCol[i]) {
count <- (count + 1)
if (count == 5) {
x11()
par(mfrow=c(4, 1))
count <- 1
}
title.histogram <- match.arg(title.histogram)
switch(title.histogram,
auto = titleH <- names(logCol[i]),
none = titleH <- '')
x <- as.matrix(df[ ,i])
tbl <- tb.make.table.II(x, k, breaks, right, histogram, titleH)
tmpList <- c(tmpList, list(tbl))
}
}
valCol <- logCol[logCol]
names(tmpList) <- names(valCol)
return(tmpList)
}
# User defines one factor
else {
namesdf <- names(df)
pos <- which(namesdf == by)
stopifnot(is.factor((df[[pos]])))
nF <- table(df[[pos]])
logCol <- sapply(df, is.numeric)
nHist <- length(logCol[logCol])
nDisGraph <- round((length(nF) * nHist) / 12) # 12 is the maximum easily visible
if (histogram) {
count <- 0
x11()
par(mfrow=c(4, 3))
}
for(i in 1:length(nF)) {
tmpdf <- subset(df, df[[pos]] == names(nF[i]))
logCol <- sapply(tmpdf, is.numeric)
for (j in 1:ncol(tmpdf)) {
if (logCol[j]) {
count <- (count + 1)
if (count == 13) {
x11()
par(mfrow=c(4, 3))
count <- 1
}
nameF <- names(nF[i])
nameY <- names(logCol[j])
nameFY <- paste(nameF,'.', nameY, sep="")
title.histogram <- match.arg(title.histogram)
switch(title.histogram,
auto = titleH <- nameFY,
none = titleH <- '')
x <- as.matrix(tmpdf[ ,j])
tbl <- tb.make.table.II(x, k, breaks, right, histogram, titleH)
newFY <- list(tbl)
names(newFY) <- sub(' +$', '', nameFY)
tmpList <- c(tmpList, newFY)
}
}
}
}
return(tmpList)
}
Testar função tb.table
O script abaixo possibilita testar e aprender a usar a função tb.table.
#===============================================================================
# Name : tb.table_test
# Original author: Jose Cláudio Faria
# Date (dd/mm/yy): 1/3/07 11:06:02
# Version : v24
# Aim : To learn how to use the function tb.table
#===============================================================================
# Observation : Test it line by line
#===============================================================================
# 1.Tables
# 1.1. Tables from vectors
#===============================================================================
## To debug
# mtrace.off()
# mtrace(tb.make.table.I)
# mtrace(tb.make.table.II)
# mtrace(tb.table.default)
# mtrace(tb.table.data.frame)
# Make a vector
set.seed(1)
x=rnorm(150, 5, 1)
tb.table(x, his=F)
tb.table(x)
tb.table(x, title.his='none')
tb.table(x, k=10, his=T)
#Title
tb.table(x, title.his='teste') #error!
tb.table(x, title.his='none')
tb.table(x, title.his='auto')
# Equal to above
tb.table(x, breaks='Sturges')
# Equal to above
tb.table(x, breaks='St')
tb.table(x, breaks='Scott')
# Equal to above
tb.table(x, b='Sc')
tb.table(x, breaks='FD')
# Equal to above
tb.table(x, breaks='F')
tb.table(x, breaks='F', right=T)
# Will make a error!
tb.table(x, breaks='S') #('S'turges) and ('S'cott)
tb.table(x, k=4)
tb.table(x, k=20)
# Partial
tb.table(x, start=4, end=6) # Will make error!
tb.table(x, start=4, end=6, his=F)
# Equal to above
tb.table(x, s=4, e=6, his=F)
# Partial
tb.table(x, start=4.5, end=5.5, his=F)
# Partial
tb.table(x, start=5, end=6, h=.5, his=F)
# Nonsense
tb.table(x, start=0, end=10, h=.5)
# First and last class forced (fi=0)
tb.table(x, start=1, end=9, h=1)
tb.table(x, start=1, end=10, h=2)
#===============================================================================
# 1.2. Tables from data.frames
#===============================================================================
# Make a data.frame
mdf=data.frame(X1 =rep(LETTERS[1:4], 25),
X2 =as.factor(rep(1:10, 10)),
Y1 =c(NA, NA, rnorm(96, 10, 1), NA, NA),
Y2 =rnorm(100, 60, 4),
Y3 =rnorm(100, 50, 4),
Y4 =rnorm(100, 40, 4))
tb.table(mdf)
tb.table(mdf, title.his='none')
# Equal to above
tb.table(mdf, breaks='Sturges')
# Equal to above
tb.table(mdf, breaks='St')
tb.table(mdf, breaks='Scott')
tb.table(mdf, breaks='FD')
tb.table(mdf, k=4)
tb.table(mdf, k=10)
levels(mdf$X1)
tbl = tb.table(mdf, k=5, by='X1')
length(tbl)
names(tbl)
tbl
tb.table(mdf, breaks='FD', by='X1')
# A 'big' result: X2 is a factor with 10 levels!
tb.table(mdf, breaks='FD', by='X2')
tb.table(mdf, breaks='FD', k=5, by='X2')
tb.table(iris, k=5)
tb.table(iris, k=10)
levels(iris$Species)
tbl=tb.table(iris, k=5, by='Species')
length(tbl)
names(tbl)
tbl
tb.table(iris, k=5, by='Species', right=T)
tb.table(iris, breaks='FD', by='Species')
library(MASS)
levels(Cars93$Origin)
tbl=tb.table(Cars93, k=5, by='Origin')
names(tbl)
tbl
tb.table(Cars93, breaks='FD', by='Origin')