CE-227 - Primeiro semestre de 2014
No quadro abaixo será anotado o conteúdo dado em cada aula do curso.
São indicados os Capítulos e Sessões correspondentes nas referências bibliográficas,
bem como os exercícios sugeridos.
Veja ainda depois da tabela as Atividades Complementares.
Observação sobre exercícios recomendados os exercícios indicados são compatíveis com o nível e conteúdo do curso.
Se não puder fazer todos, escolha alguns entre os indicados.
Conteúdos das Aulas
| Data | Conteúdo | Leitura | Exercícios | Tópico |
|---|---|---|---|---|
| 12/02 Qua | Teorema de Bayes: revisão, interpretações e generalização. Expressão probabilística de informação subjetiva, estimação baseada nos dados, estimação combinando informação prévia (subjetiva) e dados. Exemplo Binomial-Beta | Ver abaixo | Ver abaixo | |
| 14/02 Sex | Exemplo (cont). Inferência Bayesiana sobre parâmetro da Binomial com priori Beta. Determinação da posteriori | Ver notas de aulas Capítulos 1 e 2 | Exercícios dos Capítulos 1 e 2 das notas de aula | |
| 19/02 Qua | Discussão dos fundamentos do paradigma Bayesiano. Resumos e formas de explorar a posteriori: resumos pontuais e intervalares (intervalos de quantis e HPD). Inferência Bayesiana da distribuição Poisson-Gama | Ver notas de aulas Cap. 1 e 2 | Exercícios dos Capítulos 1 e 2 das notas de aula Ver abaixo | |
| 21/02 Sex | Discussão de possíveis soluções computacionais para especificação de prioris, resumos das posterioris incluindo intervalos HDP | Ver notas de aulas Capítulo 3 | Exercícios do Capítulo 3 das notas de aula | |
| 26/02 Qua | Avaliação semanal transferida devido a greve de ônibus. Explorando posterioris para prioris não conjugadas e que não possuem forma analítica conhecida. Métodos discutidos: (i) aproximação (Normal e Laplace) e (ii) amostragem da posteriori (aceitação/rejeição e MCMC) | Ver abaixo | ||
| 28/02 Sex | 1a Avaliação semanal (sabatina). Discussão da questão com destaque a resumos da posteriori. Especificação de Prioris: conjugadas, impróprias e "não informativas". Representações de ignorância e priori de Jeffreys. | Notas de aula, Cap. 3 | Ver abaixo | |
| 05/03 Sex | Feriado Carnaval | |||
| 07/03 Sex | 2a Avaliação semanal (sabatina). Resultados assintóticos e aproximação normal da posteriori. Algorítmo MCMC. | Notas de aula, Cap 7 | Ver abaixo | |
| 12/03 Qua | Predição bayesiana. Inferência para vetor de parâmetros. | Cap 4, Cap 6 | ||
| 14/03 Sex | Discussão sobre princípios e métodos bayesianos. Algorítmo de Gibbs. | |||
| 19/03 Qua | Princípio da verossimilhança. Histórico e contraste entre paradigmas para inferência | |||
| 21/03 Sex | 3a Avaliação semanal (sabatina). Revisão sobre métodos para obter e fazer inferências com posteriori: analítico (conjugado ou não), numérico, aproximação discreta, aproximação Gaussiana, simulação. Reparametrização e efeitos em aproximações e algoritmos de simulação. | Ver abaixo | ||
| 26/03 Qua | Priori's definidas por misturas, prioris de referências e para modelos de locação ou escala. Resumos pontuais e funções perda. Comentários sobre a questão da 3a avaliação. | Ver abaixo | ||
| 29/03 Sex | Discussão sobre a questão de 3a avaliação semanal | Ver abaixo | ||
| 02/03 Qua | Computação da questão de 3a avaliação semanal. Análise Bayesiana de dados: modelos linerares (Gaussianos). Construção do algorítmo de Gibbs | Ver abaixo | ||
| 04/04 Sex | Extensões e fundamentos do algorítmo de Gibbs. Gibbs metropolizado. Predição. Introdução do JAGS e rjags | Ver abaixo | ||
| 09/04 Qua | Inferência Bayesiana em modelos lineares generalizados (ex distribuição de Poisson). Introdução aos modelos de efeitos aleatórios - comparações com modelos de efeitos fixos e verossimilhança | Ver abaixo | ||
| 11/04 Sex | Modelos de efeitos aleatórios/hierárquicos e inferência Bayesiana. Algorítmos de inferência e INLA | Ver abaixo | ||
| 16/04 Qua | Avaliação semanal. Discussão da avaliação sobre modelos declarados em JAGS. Distribuições marginais 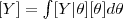 e interpretação. Exemplo: caso Poisson e interpretação. Exemplo: caso Poisson | Ver abaixo | ||
| 23/04 Qua | sem aula expositiva. Preparar e discutir atividades propostas na última aula para apresentação na próxima aula. | |||
| 25/04 Sex | Avaliação: apresentação das análises de dados correspondentes aos modelos da quarta avaliação semanal | |||
| 30/04 Sex | continuação das apresentações e discussões | |||
| 07/05 Qua | Modelos para efeitos aleatórios dependentes. Estrutura do modelo, comparação com outras estratégias/modelos. Exemplo: série temporal para dados binários. | Ver abaixo | ||
| 09/05 Sex | Fundamentos do INLA | Apresentação | ||
| 14/05 Sex | Fundamentos do INLA - II - Comentários sobre a (excelente!) apresentação de Gianluca Baio | A apresentação |
12/02
- Procurar materiais introdutórios sobre inferência Bayesiana na web, livros, etc
- Escrever um programa que encontre os parâmetros da distribuição Beta a partir de estimativas pontuais, intervalos e respectiva probabilidade (como visto na aula)
- Investigar como foram encontrados os parâmetros na distribuição que combina dados e informações subjetivas mostrada em aula.
Seguem alguns resultados obtidos com meu código para encontrar os parâmetros da priori beta a partir de um valor que considera-se ser a moda, uma faixa de valores e uma probabilidade associada a esta faixa
| Opinião | Parâmetros da Priori | Parâmetros da Posteriori | Credibilidade (95%) | Credibilidade (95%) | ||||
|---|---|---|---|---|---|---|---|---|
| Escolha | Moda | Intervalo | alfa | beta | alfa* | beta* | quantis | HPD |
| 1 | 0.20 | [0.15 , 0.35] | 3.24 | 9.95 | 27.24 | 65.95 | [0.20 , 0.39] | [0.20 , 0.38] |
| 2 | 0.40 | [0.10 , 0.70] | 3.84 | 5.25 | 27.84 | 61.25 | [0.22 , 0.41] | [0.22 , 0.41] |
| 3 | 0.40 | [0.38 , 0.42] | 33.67 | 50.00 | 57.67 | 106.00 | [0.28 , 0.43] | [0.28 , 0.43] |
| 4 | 0.60 | [0.55 , 0.65] | 74.59 | 50.00 | 98.50 | 106.00 | [0.41 , 0.55] | [0.41 , 0.55] |
| 5 | 0.50 | [0.20 , 0.80] | 2.06 | 2.06 | 26.06 | 58.06 | [0.22 , 0.41] | [0.21 , 0.41] |
| 6 | 0.65 | [0.50 , 0.90] | 21.01 | 11.78 | 45.01 | 67.78 | [0.31 , 0.49] | [0.31 , 0.49] |
19/02
- Considere o exemplo visto em aula (lembrando que consideramos n=80 e y=24) e a sua escolha de priori e mostre como obter resumos pontuais da posteriori do modelo Binomial-Beta de duas formas distintas:
- usando resultados analíticos da distribuição,
- através de algum algorítmo computacional.
- Mostrar como obter intervalos de quantis e HPD para posteriori do modelo Binomial-Beta. Escrever rotinas computacionais e obter resultados para o exemplo de aula.
- Repetir anteriores para o modelo Poisson-Gama
26/02
- Obter amostras das posterioris nos modelo Binomial-Beta e Poisson-Gama. Extrair resultados e comparar com os analíticos obtidos anteriormente.
- Obter a aproximação de Normal para as posterioris destes dois modelos
- Escrever um algoritmo MCMC para obter amostras destes dois modelos (supondo - artificialmente - que a posteriori não é de forma conhecida).
28/02
- Refazer a questão da 1a avaliação.
- Encontrar a aproximação de Normal para a posteriori da questão da 1a avaliação. Fazer gráfico sobrepondo as distribuições, comparar resumos descritivos das distribuições. Avaliar a aproximação.
- Reconsidere a questão da 1a avaliação utilizando o parâmetro de precisão
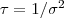 . Encontre a priori correspondente à definida (por transformação de variáveis).
. Encontre a priori correspondente à definida (por transformação de variáveis). - Mostre que para questão da 1a avaliação a distribuição gama inversa é conjugada. Idem para distribuição gama para o parâmetro de precisão.
- Considere agora a distribuição indexada para parâmetro (reparametrização)
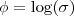 . Encontre a priori equivalente na definida na avaliação, encontre a distribuição a posteriori e a aproximação da Laplace correspondente. Sobreponha em um gráfico e discuta.
. Encontre a priori equivalente na definida na avaliação, encontre a distribuição a posteriori e a aproximação da Laplace correspondente. Sobreponha em um gráfico e discuta. - Monte um algoritmo MCMC para amostrar de algumas das posterioris da avaliação ou itens anteriores. Sobreponha em um gráfico a densidade obtida e a estimada pelas amostras MCMC. Transforme os valores simulados de acordo com as transformações dos itens anteriores e novamente compare graficamente as posterioris obtidas analiticamente e pelas simulações.
07/03
- Obter a aproximação normal para os exemplos vistos até agora no curso (modelos Poisson, Geométrico, Binomial Negativo, Binomial, Expoencial, Gamma, …)
- Montar um algorítmo MCMC para os exemplos vistos até agora no curso (modelos Poisson, Geométrico, Binomial Negativo, Binomial, Exponencial, Gamma, …)
21/03
- Considerar o problema da 3a avaliação semanal. Resolver analicamente e por métodos numéricos.
26/03
- Considere o modelo binomial e uma amostra com n=15 e y=6. Obtenha os gráficos da "trinca" priori, verossimilhança e posteriori nos seguintes casos:
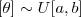
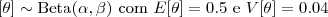
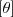 : priori de Jeffrey's
: priori de Jeffrey's
29/03
- Implementar algorítmos de inferência para o problema da 3a avaliação semanal. Fazer gráficos das quantidades e funções de interesse.
02/04
- Obter as expressões analíticas das posterioris conjunta, marginais e condicionais
do modelo de regressão linear com as prioris de referência.
- Estender o código a seguir (visto em aula) para comparar os resultados analíticos com os obtidos pelo amostrador de Gibbs. Incluir na comparação as distribuições e resumos pontuais e intervalares.
- Generalizar os resultados analíticos e códigos para priori Normal-InversaGamma
## simulando dados
x <- sort(runif(20, 0, 20))
y <- 2 + 0.25*x + rnorm(20, m=0, sd=2)
## visualizando dados e reta de regressão "usual"
plot(x,y)
reg <- lm(y ~ x)
abline(reg)
## Código ("ingênuo") para amostrador de Gibbs
reg.lin.GIBBS <- function(y, x, Nsim, iniBeta){
require(MASS)
n <- length(y)
X <- cbind(1, x)
XX <- crossprod(X)
bhat <- solve(XX,crossprod(X,y))
##
sim <- matrix(0, nrow = Nsim, ncol=3)
sim[1,1:2] <- iniBeta
sim[1, 3] <- 1/rgamma(1, shape=(n-2)/2, scale=2/crossprod(y-X%*%sim[1,1:2]))
for(i in 2:Nsim){
sim[i, 3] <- 1/rgamma(1, shape=(n-2)/2, scale=2/crossprod(y-X%*%sim[(i-1),1:2]))
sim[i,1:2] <- mvrnorm(n=1, mu=bhat, Sigma = solve(XX)*sim[i, 3])
}
return(sim)
}
## Obtendo amostras
rlG0 <- reg.lin.GIBBS(y=y, x=x, Nsim=15000, iniBeta=c(0,0.6))
## Burn-in (3000) e thining (1/10)
rlG1 <- rlG0[-(1:3000),]
rlG2 <- rlG1[10*(1:1200),]
## comparando estimativas "usuais" e médias das posterioris
c(coef(reg), summary(reg)$sigma^2)
apply(rlG2, 2, mean)
## traços e posterioris (por simulação) com indicação das estimativas "usuais"
par(mfrow=c(2,3))
plot(rlG2[,1], type="l")
plot(rlG2[,2], type="l")
plot(rlG2[,3], type="l")
plot(density(rlG2[,1])); abline(v=coef(reg)[1])
plot(density(rlG2[,2])); abline(v=coef(reg)[2])
plot(density(rlG2[,3])); abline(v=summary(reg)$sigma^2)
04/04
- Estender os códigos anteriores para incluir a predição de novos valores
#### código agora incluindo predições
reg.lin.GIBBS <- function(y, x, Nsim, iniBeta, x.pred = NULL){
require(MASS)
n <- length(y)
X <- cbind(1, x)
XX <- crossprod(X)
bhat <- solve(XX,crossprod(X,y))
##
NC <- ncol(X) + 1
if(!is.null(x.pred)){
Xpred <- cbind(1, x.pred)
NC <- NC + nrow(Xpred)
}
sim <- matrix(0, nrow = Nsim, ncol=NC)
sim[1,1:2] <- iniBeta
sim[1, 3] <- 1/rgamma(1, shape=(n-2)/2, scale=2/crossprod(y-X%*%sim[1,1:2]))
##
for(i in 2:Nsim){
sim[i, 3] <- 1/rgamma(1, shape=(n-2)/2, scale=2/crossprod(y-X%*%sim[(i-1),1:2]))
sim[i,1:2] <- mvrnorm(n=1, mu=bhat, Sigma = solve(XX)*sim[i, 3])
if(!is.null(x.pred))
sim[i,-(1:3)] <- rnorm(nrow(Xpred), m=Xpred%*%sim[i,1:2], sd=sqrt(sim[i,3]))
}
return(sim)
}
## agora com predição
rlG0 <- reg.lin.GIBBS(y=y, x=x, Nsim=15000, iniBeta=c(0,0.6), x.pred=1:20)
dim(rlG0)
rlG1 <- rlG0[-(1:3000),]
dim(rlG1)
rlG2 <- rlG1[10*(1:1200),]
dim(rlG2)
rbind(dim(rlG0), dim(rlG1), dim(rlG2))
reg
summary(reg)$sigma^2
par(mfrow=c(4,5))
apply(rlG2[,-(1:3)], 2, function(x) plot(density(x),type="l"))
par(mfrow=c(1,1))
y.pred <- apply(rlG2[,-(1:3)], 2, mean)
plot(y ~ x)
abline(reg)
lines(1:20, y.pred, col=2)
#
y.pred <- apply(rlG1[,-(1:3)], 2, mean)
plot(y ~ x)
abline(reg)
lines(1:20, y.pred, col=2)
- Incluir bandas de predição usuais e bayesianas no gráfico
- Generalizar código para família de priori Normal-GammaInversa
- Verificar o efeito/sensitividade à especificação das prioris
Exemplos JAGS/rjags
- Média e variância (precisão) da distribuição normal
n <- 20 x <- rnorm(n, 70, 5) #write.table(x, # file = 'normal.data', # row.names = FALSE, # col.names = FALSE) cat( "model { for (i in 1:n){ x[i] ~ dnorm(mu, tau) } mu ~ dnorm(0, 0.0001) tau <- pow(sigma, -2) sigma ~ dunif(0, 100) }", file="normal.modelo" ) require(rjags) ## OBS: valores iniciais são dispensáveis neste exemplo inis <- list(list(mu=10, sigma=2), list(mu=50, sigma=5), list(mu=70, sigma=10)) jags <- jags.model('normal.modelo', data = list('x' = x, 'n' = n), n.chains = 3, inits = inis, n.adapt = 100) #update(jags, 1000) #sam <- jags.samples(jags, c('mu', 'tau'), 1000) sam <- coda.samples(jags, c('mu', 'tau'), n.iter=10000, thin=10) par(mfrow=c(2,2)) plot(sam) str(sam) summary(sam) HPDinterval(sam) - regressão linear simples
cat( "model { for (i in 1:n){ y[i] ~ dnorm(mu[i], tau) mu[i] <- b0 + b1 * x[i] } b0 ~ dnorm(0, .0001) b1 ~ dnorm(0, .0001) tau <- pow(sigma, -2) sigma ~ dunif(0, 100) }", file="reglin.modelo") ## alternativa: ## tau ~dgamma(0.001, 0.001) ## sigma2 <- 1/tau n <- 20 x <- sort(runif(n, 0, 20)) epsilon <- rnorm(n, 0, 2.5) y <- 2 + 0.5*x + epsilon #write.table(data.frame(X = x, Y = y, Epsilon = epsilon), # file = 'reglin.dados', # row.names = FALSE, # col.names = TRUE) require(rjags) # require(R2jags) # require(dclone) inis <- list(list(b0=0, b1=1, sigma=1), # list(b0=1, b1=0.5, sigma=2), list(b0=2, b1=0.1, sigma=5)) jags <- jags.model('reglin.modelo', data = list('x' = x, 'y' = y, 'n' = n), n.chains = 2, # inits=inits, n.adapt = 100) #update(jags, 1000) class(jags) sam <- jags.samples(jags, c('b0', 'b1', 'sigma'), 1000) class(sam) sam <- coda.samples(jags, c('b0', 'b1', 'sigma'), 1000) class(sam) str(sam) plot(sam) sam <- coda.samples(jags, c('b0', 'b1', 'sigma', "y"), 1000) str(sam) int <- HPDinterval(sam) str(int) ## complementar com gráficos, resumos, inferências de interesse, etc
09/04
- Fazer uma análise Bayesiana de uma regressão de Poisson e comparar com resultados de um MLG (modelo linear generalizado) usual. Utilize um conjunto de dados qualquer da literatura (sugestão: conjunto
galado pacote "faraway") - Use os dados do artigo Confiabilidade e Precisão na Estimação de Médias de Singer et al na Revista Brasileira de Estatística, v73, n. 236, jan./jun 2012 e:
- ajuste um modelo "ingênuo" de média e resíduo
- ajuste um modelo de média, efeito de local e resíduo (medidas repetidas com efeitos fixos)
- ajuste um modelo de efeitos aleatórias
- ajuste um modelo Bayesiano.
Compare e discuta os resultados.
11/04
##
## Dados simulados do modelo:
## Y_{ij} \sim N(\mu_{i}, \sigma^2_y)
## mu_{i} = theta + b_{i}
## b_{i} \sim N(0, \sigma^2_b)
## que, por ser normal (com ligação identidade)
## pode ser escrito por:
## Y_{ij} = \beta_0 + b_{i} + \epsilon_{ij}
##
Ngr <- 25
Nobs <- 10
set.seed(12)
sim <- data.frame(id = Ngr*Nobs,
gr = rep(1:Ngr, each=Nobs),
bs = rep(rnorm(Ngr, m=0, sd=10), each=Nobs),
eps = rnorm(Ngr*Nobs, m=0, sd=4)
)
sim <- transform(sim, y = 100 + bs + eps)
sim
## estimativas "naive"
resumo <- function(x) c(media=mean(x), var=var(x), sd=sd(x), CV=100*sd(x)/mean(x))
(sim.res <- aggregate(y~gr, FUN=resumo, data=sim))
var(sim.res$y[,1])
mean(sim.res$y[,2])
mean(sim$y)
## Modelo de efeitos aleatórios
require(lme4)
fit.lme <- lmer(y ~ 1|gr, data=sim)
summary(fit.lme)
ranef(fit.lme)
coef(fit.lme)$gr - fixef(fit.lme)
print(VarCorr(fit.lme), comp="Variance")
## JAGS
require(rjags)
sim.lst <- as.list(sim[c("gr","y")])
sim.lst$N <- nrow(sim)
sim.lst$Ngr <- length(unique(sim$gr))
mean(sim.lst$y)
cat("model{
for(j in 1:N){
y[j] ~ dnorm(mu[gr[j]], tau.e)
}
for(i in 1:Ngr){
mu[i] ~ dnorm(theta, tau.b)
}
theta ~ dnorm(0, 1.0E-6)
tau.b ~ dgamma(0.001, 0.001)
sigma2.b <- 1/tau.b
tau.e ~ dgamma(0.001, 0.001)
sigma2.e <- 1/tau.e
cci <- sigma2.e/(sigma2.e+sigma2.b)
}", file="sim.jags")
sim.jags <- jags.model(file="sim.jags", data=sim.lst, n.chains=3, n.adapt=1000)
## inits = ...
fit.jags <- coda.samples(sim.jags, c("theta", "sigma2.b", "sigma2.e", "cci"), 10000, thin=10)
summary(fit.jags)
plot(fit.jags)
##
require(INLA)
fit.inla <- inla(y ~ f(gr) , family="gaussian", data=sim)
summary(fit.inla)
sqrt(1/fit.inla$summary.hyperpar[,1])
16/04
- (Individual ou em duplas) Encontre um conjunto de dados para cada um dos casos na avaliação semanal de 16/04. Proceda análises não Bayesianas e Bayesianas e discuta os resultados. Trazer scripts para discussão na aula de 23/04 e apresentação em 25/04.
- Considere o modelo de verossimilhança
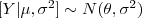 e a priori
e a priori 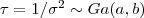 . Mostre como obter a densidade:
. Mostre como obter a densidade:
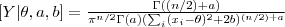 .
.
Como este resultado pode ser interpretado?
07/05
require(INLA) ## ## Visualizado dados ## data(Tokyo) head(Tokyo) plot(y ~ time, data=Tokyo) ## colocando na forma de proporção de dias com chuva plot(y/2 ~ time, data=Tokyo) ## ## 1. Modelo "Nulo": só intercepto ## estimando a probabilidade de chuva como uma constante: fit.glm <- glm(cbind(y, n-y) ~ 1, family=binomial, data=Tokyo) abline(h=exp(coef(fit.glm))/(1+exp(coef(fit.glm))), col=2, lty=3, lwd=3) ## como, como este modelo, todos os valores preditos são iguais bastaria fazer: abline(h=fitted(fit.glm)[1], col=2, lty=3, lwd=3) ## ## 2. Modelo com probabilidades variando no tempo (variável/processo latente) ## modelando o logito(probabilidade) como um efeito aleatório correlacionado no tempo ## segundo um "random walk" cíclico de ordm 2 modelo = y ~ 0 + f(time, model="rw2", cyclic=T, param=c(1, 0.0001)) fit <- inla(modelo, data=Tokyo, family="binomial", Ntrials=n, control.predictor=list(compute=TRUE)) ## names(fit) head(fit$summary.fitted.values) ## sobrepondo ao gráfico dos dados (moda, mediana e médi são praticamente indistinguíveis with(fit, matlines(summary.fitted.values[,c(1,3:6)], lty=c(1,2,2,2,3), col=1)) ## ## 3. Agora o mesmo modelo nulo anterior porém jusado pelo INLA ## modelo0 = y ~ 1 fit0 <- inla(modelo0, data=Tokyo, family="binomial", Ntrials=n, control.predictor=list(compute=TRUE)) summary(fit0) fit0$summary.fitted.values with(fit0, matlines(summary.fitted.values[,c(1,3:6)], lty=c(1,2,2,2,3), col=2)) ## ## 4. Modelando usando GAM (generalised additive model) ## require(mgcv) fit.gam <- gam(cbind(y, n-y) ~ s(time), family=binomial, data=Tokyo) names(fit.gam) fitted(fit.gam, se=T) pred.gam <- predict(fit.gam, type="response", se=T, newdata=Tokyo["time"]) names(pred.gam) with(pred.gam, matlines(cbind(fit, fit+2*se.fit, fit-2*se.fit), lty=c(1,2,2), col=4))